Биохимия, биотехнология и диагностика
УДК 619.616.993.192.6
DOI:10.12737/16663
Поступила в редакцию16.10.2015
Принята в печать 24.11.2015
Скачать статью в PDF формате
English version
Днк-диагностика анаплазмоза крупного рогатого скота
Самуйленко А. Я.1, Гулюкин М. И. 2, Василевич Ф. И. 3, Ковальчук С. Н. 4, Глазко Т. Т. 4, 5, Бабий А. В.4, Архипов А. В.4, Косовский Г. Ю.4
1 Всероссийский научно-исследовательский и технологический институт биологической промышленности (ФГБНУ ВНИТИБП)
141142, Московская область, Щелковский р-н, пос. Биокомбината
2 Всероссийский научно-исследовательский институт экспериментальной ветеринарии
им. Я. Р. Коваленко (ФГБНУ ВИЭВ)
109428, Москва, Рязанский проспект, к. 1, д. 24
3 Московская государственная академия ветеринарной медицины и биотехнологии
им. К. И. Скрябина
109472, Москва, ул. Академика Скрябина, д. 23
4 Центр экспериментальной эмбриологии и репродуктивных биотехнологий (ФГБНУ ЦЭЭРБ)127422, Москва, ул. Костякова, д. 12, стр. 4, e-mail: info-ceerb@mail.ru
Реферат
Цель исследования – разработка метода ДНК-диагностики анаплазмоза крупного рогатого скота.
Материалы и методы. Пробы крови отбирали из хвостовой вены с использованием ЭДТА в качестве антикоагулянта. ДНК выделяли с помощью набора Sorb-M. Для анализа гена msp4 были использованы соответствующие последовательности, принадлежащие разным изолятам Anaplasma marginale. Выявление консервативных участков последовательностей для подбора праймеров проводили с помощью сервера СlustalW2. Видоспецифичность праймеров проверяли с использованием алгоритма BLASTN. Результаты полимеразной цепной реакции (ПЦР) оценивали методом электрофореза в 2%-ном агарозном геле. Электрофорез проводили в течение 40 минут при напряженности поля 5 В/см. Полученные в результате ПЦР фрагменты гена msp4 были очищены, лигированы и клонированы в клетках E. coli. Трансформацию проводили методом теплового шока. Поиск колоний E. coli DH5α, содержащих плазмиду pGEM-msp4, проводили методом ПЦР с использованием стандартных праймеров M13 с последующим анализом результатов ПЦР методом электрофореза. Целевые колонии наращивали в течение ночи при 37 °С в 2 мл среды LB, содержащей ампициллин в концентрации 100 мкг/мл. Секвенирование полученных плазмид pGEM-msp4 осуществляли по методу Сэнгера и генетического анализатора Applied Biosystems 3130.
Результаты и обсуждение. Описаны разработка и апробация праймеров к гену msp4 Anaplasma marginale для ДНК-диагностики анаплазмоза крупного рогатого скота методом ПЦР. Чувствительность ПЦР с использованием этих праймеров позволяет выявить 100 и больше копий гена. Проведенные испытания свидетельствуют о 100%-ной повторяемости и воспроизводимости данного метода.
Ключевые слова: Anaplasma marginale, крупный рогатый скот, диагностика, ПЦР.
Введение
Анаплазмоз крупного рогатого скота (КРС) – трансмиссивное инфекционное заболевание, вызываемое риккетсиями рода Anaplasma (отряд Riсkettsiales, семейство Anaplasmatacеa). Анаплазмоз КРС широко распространен во всем мире и приводит к значительным экономическим потерям вследствие уменьшения мясо-молочной продуктивности скота, ущерба от недополучения молодняка и гибели животных. Основным возбудителем анаплазмоза у КРС являются риккетсии Anaplasma. А. marginale – облигатный внутриклеточный паразит, поражающий эритроциты. Источником возбудителя являются инфицированные и больные животные, переносчиками – около 20 видов клещей, а также кровососущие насекомые. Возможна механическая передача возбудителей от зараженных животных к здоровым через нестерильные инструменты при проведении обезроживания, кастрации, вакцинации, при взятии крови и других зоотехнических мероприятий [1, 5, 10].
Анаплазмоз КРС, вызванный A. marginale, зарегистрирован во многих тропических и субтропических странах. Он распространен фактически по всей территории США и Канады, в некоторых странах Европы. Анаплазмоз КРС является эндемичным для Мексики, Центральной и Южной Америки, Карибских островов, Африки и Азии. В Европе А. marginale встречается главным образом в Средиземноморских странах [7]. Это заболевание регистрируют в Украине, Белоруссии, Молдавии, Казахстане, государствах Средней Азии и Закавказья [2].
Согласно ветеринарной отчетности в РФ неблагополучными по анаплазмозу являются субъекты Центрального, Северо-Западного и Приволжского ФО [4].
На сегодняшний день для диагностики анаплазмоза применяют микроскопические и серологические методы исследования, однако их чувствительность и специфичность недостаточно высоки. Результаты микроскопических исследований мазков крови ненадежны на ранних стадиях инфицирования и в случаях заболеваний, сопровождающихся тяжелой формой анемии [12]. Серологические методы, основанные на использовании антител к антигенам возбудителя анаплазмоза, имеют недостаточно высокую чувствительность [13] и не позволяют дифференцировать А. marginale от других видов анаплазм [14, 16]. Наиболее оперативную и точную диагностическую информацию позволяет получить выявление ДНК возбудителей анаплазмоза в крови КРС с помощью полимеразной цепной реакции. Преимуществом ПЦР-диагностики является высокая чувствительность и специфичность – она позволяет обнаружить возбудителя на самых ранних стадиях заболевания, в том числе и во время латентной стадии, и надежно дифференцировать анаплазмоз от ряда сходных по клиническим проявлениям заболеваний.
Целью настоящих исследований была разработка способа выявления ДНК A. marginale в периферической крови крупного рогатого скота методом ПЦР.
Материалы и методы
Выделение ДНК. Пробы крови отбирали из хвостовой вены стерильными катетерами с использованием ЭДТА в качестве антикоагулянта. ДНК выделяли с помощью набора Sorb-M (Синтол, Россия) согласно рекомендациям производителя.
Конструирование праймеров для ПЦР. Для анализа гена msp4 были использованы соответствующие последовательности, принадлежащие разным изолятам A. marginale доступные в базе данных GeneBank (http://www.ncbi.nlm.nih.gov/genbank/). Выявление консервативных участков последовательностей для подбора праймеров проводили с помощью сервера СlustalW2 (http://www.ebi.ac.uk/Tools/msa/clustalw2/). Видоспецифичность праймеров была проверена с использованием алгоритма BLASTN (http://blast.ncbi.nlm.nih.gov/Blast.cgi). Визуализацию результатов проводили с помощью программы GeneDoc.
Проведение ПЦР. Реакцию амплификации проводили в 10 мкл смеси для ПЦР, содержащей 1×буфер для амплификации, 1 а. е. HS Таq ДНК-полимеразы (Евроген, Россия), 200 мкМ нуклеозидтрифосфатов, 0,2 мкМ праймера MSP4-F: 5′- AAGGGGGAGTAATGGGAGGTA -3′, 0,2 мкМ праймера MSP4-R: 5′-GGCACACTCACATCAATC -3′, 3 мкл ДНК. ПЦР проводили при следующих условиях: начальная денатурация в течение 3 мин при 95 °С; 45 циклов (15 с при 95 °С, 15 с при 58 °С, 15 с при 72 °С); финальная элонгация при 72 °С в течение 3 мин. ПЦР проводили в термоциклерах LightCycler 96 System (Roche, Швейцария) и Nyx Technik Amplitronyx 6 (США).
Электрофорез. Результаты ПЦР оценивали методом электрофореза в 2%-ном агарозном геле. Электрофорез проводили в 1xTAE буфере, содержащем раствор бромистого этидия в концентрации 0,5 мкг/мл. Электрофорез проводили в течение 40 мин при напряженности поля 5 В/см. Визуализацию результатов проводили с помощью трансиллюминатора ECX-F20.M (Vilber Lourmat, Франция).
Получение контрольной плазмиды pGEM-msp4. Полученные в результате ПЦР фрагменты гена msp4 были очищены с помощью набора GeneJET PCR Purification Kit (Life technologies, USA), лигированы в вектор pGEM-T (Promega, США) и клонированы в клетках E. coli DH5α. Трансформацию проводили методом теплового шока. Поиск колоний E. coli DH5α, содержащих плазмиду pGEM-msp4, проводили методом ПЦР с использованием стандартных праймеров M13 с последующим анализом результатов ПЦР методом электрофореза в 2%-ном агарозном геле. Целевые колонии наращивали в течение ночи при 37 °С в 2 мл среды LB, содержащей ампициллин в концентрации 100 мкг/мл. Очистку плазмидной ДНК проводили с использованием набора GeneJET Miniprep Kit (Thermo Fisher Scientific, США), оценку концентрации плазмидной ДНК – с помощью набора PicoGreen® dsDNA Assay Kit (Thermo Fisher Scientific, США) с использованием флуориметра QuantiFluor-ST (Promega, США).
Секвенирование. Секвенирование полученных плазмид pGEM-msp4 осуществляли по методу Сэнгера с использованием набора ABI Prism Big Dye Terminator 3.1 Cycle Sequencing Kit (Applied Biosystems, США) согласно рекомендациям изготовителя и генетического анализатора Applied Biosystems 3130 (Life Technologies, США).
Результаты и обсуждение
Своевременная диагностика играет решающую роль в предотвращении распространения инфекционных заболеваний. ПЦР является широко используемым методом диагностики инфекционных заболеваний благодаря его высокой чувствительности, специфичности, воспроизводимости и относительной быстроте проведения анализа. Согласно рекомендациям Всемирной организации здравоохранения животных (OIE, the World Organisation for Animal Health), изложенным в «Руководстве по диагностическим тестам и вакцинам для наземных животных 2015 г.» (http://www.oie.int/en/international-standard-setting/terrestrial-manual/access-online/), ПЦР-диагностику A. marginale следует применять перед каждым перемещением животного на новое место и для подтверждения диагноза. На сегодняшний день описаны разные методы ПЦР для детекции A. marginale [6, 8, 9, 11, 15], однако ни один из них не прошел полную валидацию.
В данном исследовании были разработаны праймеры для амплификации фрагмента гена msp4 методом ПЦР с целью выявления А. marginale в крови КРС. Msp4 является иммунодоминантным белком наружной мембраны [7]. С целью подбора праймеров для msp4-ПЦР были проанализированы имеющиеся в международной базе данных Генбанк последовательности гена msp4 А. marginale. Сравнительный анализ последовательностей msp4 показал высокую степень их идентичности – 98–100 %. Прямой и обратный праймеры были подобраны на участки гена msp4, имеющие идентичность 100 % у всех известных изолятов А. marginale.
Видоспецифичность праймеров MSP4-F и MSP4-R была проверена in silico путем сравнения их последовательностей с геномными библиотеками с использованием программы BLASTN. В результате 100 % покрытия и идентичности последовательностей праймеров было получено со всеми последовательностями гена msp4 A. marginale, имеющимися в базе данных Генбанк. Полная комплементарность праймеров к участкам генома каких-либо нецелевых организмов не выявлена.
Подобранные праймеры MSP4-F и MSP4-R были использованы для проведения ПЦР, в которой в качестве матрицы использовалась ДНК коровы, инфицированной A. marginale по результатам проведенного нами ранее пиросеквенирования фрагментов генома голштинизированного скота [3]. В результате ПЦР был получен фрагмент ДНК длиной 157 п. н. Для подтверждения амплификации фрагмента целевого гена полученный фрагмент ДНК был лигирован в вектор pGEM-T (Promega, США) и клонирован в клетках E. coli DH5α. Выделенные плазмиды были секвенированы по методу Сэнгера. Анализ полученных нуклеотидных последовательностей с помощью программы BLAST показал, что они имеют 99–100 % идентичности с фрагментом гена msp4 разных изолятов A. marginale.
Для испытания аналитической специфичности праймеров использовали образцы ДНК КРС, содержащие ДНК бактерий-симбионтов или патогенов – Sanguibacter keddieii, Propionibacterium acnes, Pseudomonas aeruginosa, Stenotrophomonas maltophilia, а также образцы ДНК овец, зараженных риккетсиями A. ovis. Анализ результатов ПЦР методом электрофореза в 2%-ном агарозном геле показал отсутствие характерного для A. marginale фрагмента ДНК длиной 157 п. н., что свидетельствует об аналитической специфичности ПЦР с праймерами MSP4-F и MSP4-R.
Для определения аналитической чувствительности ПЦР была выполнена серия десятикратных разведений плазмиды pGEM-msp4, содержащей фрагмент гена msp4 длиной 157 п. н., и получены образцы, содержащие 107−100 копий гена msp4. В результате чувствительность ПЦР позволила выявить 100 копий гена msp4 и выше (рис. 1).
Для определения повторяемости и воспроизводимости метода образцы, содержащие 102−107 копий гена msp4, были проанализированы в шести повторностях. При испытании воспроизводимости амплификация проводилась разными специалистами в разные дни и на разных амплификаторах. В результате воспроизводимость и повторяемость метода составила 100 %.

Рис. 1. Результат амплификации 1000 копий (1–3), 100 копий (4–6), 10 копий (7–9) и 1 копии (10–12) плазмиды pGEM-msp4 (M – маркер молекулярного веса MWM-100RL)
Разработанный нами метод выявления A. marginale был апробирован на образцах ДНК, выделенной из цельной крови коров. Исследование проводили в трех повторностях. В качестве внутреннего положительного контроля, позволяющего оценить корректность постановки реакции и отсутствие ингибиторов, использовали плазмиду pGEM-msp4. В качестве отрицательного контроля использовали образец, не содержащий ДНК. Результаты исследования показаны на рисунке 2.
Рис. 2. 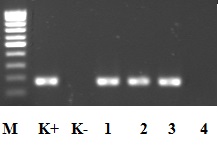 Результат амплификации ДНК, выделенной из цельной крови инфицированных (1–3) и неинфицированных (4) животных, с помощью праймеров MSP4-F и MSP4-R:
Результат амплификации ДНК, выделенной из цельной крови инфицированных (1–3) и неинфицированных (4) животных, с помощью праймеров MSP4-F и MSP4-R:
«К+» − положительный контроль; «K−» − отрицательный контроль, M − маркер молекулярной массы
Заключение
Таким образом, нами разработан и апробирован метод ДНК-диагностики анаплазмоза крупного рогатого скота. Чувствительность метода позволяет выявить 100 копий гена msp4 A. marginale и выше. Проведенные испытания свидетельствуют о 100%-ной повторяемости и воспроизводимости данного метода.
Литература
1. Василевич Ф. И., Белименко В. В., Гулюкин М. И., Георгиу Х. Практическое руководство по борьбе с кровепаразитарными болезнями домашних животных. – Москва: ЗооВетКнига, 2015. – 86 с.
2. Георгиу Х., Белименко В. В. Современные лабораторные методы диагностики анаплазмоза крупного рогатого скота и овец // РВЖ. СХЖ. – 2014. – № 3. – С. 31–32.
3. Глазко В. И., Косовский Г. Ю., Ковальчук С. Н. и др. Инвертированный повтор микросателлита (AGC)6G фланкирует районы ДНК с участками гомологии к ретротранспозонам в геноме крупного рогатого скота // Инновационные технологии в медицине. – 2014. – № 2, Т. 3. – С. 63–79.
4. Гулюкин М. И., Заблоцкий В. Т., Белименко В. В. Мониторинг эпизоотической ситуации по протозойным кровепаразитарным болезням домашних животных в Российской Федерации (2007–2012) // РВЖ. СХЖ. – 2013. – № 2. – С. 36–40.
5. Гулюкин М. И., Заблоцкий В. Т., Белименко В. В. и др. Кровепаразитарные болезни домашних животных (Атлас). – Москва: ЗооВетКнига, 2013. – 86 с.
6. Carelli G., Decaro N., Lorusso A. et al. Detection and quantification of Anaplasma marginale DNA in blood samples of cattle by real-time PCR // Vet. Microbiol. – 2007. – Vol. 124. – P. 107–114.
7. de la Fuente J., Lew A., Lutz H. et al.Genetic diversity of Anaplasma species major surface proteins and implications for anaplasmosis serodiagnosis and vaccine development // Anim. Health Res. Rev. – 2005. – Vol. 6. – P. 75–89.
8. de la Fuente J., Van Den Bussche R. A., Kocan K. M. Molecular phylogeny and biogeography of North American isolates of Anaplasma marginale (Rickettsiaceae: Ehrlichieae) // Vet Parasitol. – 2001. – Vol. 97. – P. 65–76.
9. Fyumagwa R. D., Simmler P., Meli M. L. et al. Prevalence of Anaplasma marginale in different tick species from Ngorongoro Crater, Tanzania // Vet Parasitol. – 2009. – Vol. 161, № 1–2. – P. 154–157.
10. Kocan K. M., de la Fuente J., Blouin E. F. et al. The natural history of Anaplasma marginale // Vet. Parasitol. – 2010. – Vol. 167. – P. 95–107.
11. Potgieter F. T., Stoltsz W. H. Anaplasmosis // Infectious Diseases of Livestock With Special Reference to Southern Africa. – 1994. – P. 408–430.
12. Reinbold J. B., Coetzee J. F., Sirigireddy K. R., Ganta R. R. Detection of Anaplasma marginale and A. phagocytophilum in Bovine Peripheral Blood Samples by Duplex Real–Time Reverse Transcriptase PCR Assay // Journal of clinical microbiology. – 2010. – Vol. 48, № 7. – P. 2424–2432.
13. Strik N. I., Alleman A. R., Barbet A. F. et al. Characterization of Anaplasma phagocytophilum major surface protein 5 and the extent of its cross-reactivity with A. marginale // Clin. Vaccine Immunol. – 2007. – Vol. 14. – P. 262–268.
14. Torina A., Agnone A., Blanda V. et al. Development and validation of two PCR tests for the detection of and differentiation between Anaplasma ovis and Anaplasma marginale // Ticks Tick Borne Dis. – 2012. – Vol. 3, № 5–6. – P. 283–287.
16.Visser E. S., McGuire T. C., Palmer G. H. et al. The Anaplasma marginale msp5 gene encodes a 19-kilodalton protein conserved in all recognized Anaplasma species // Infect Immun. – 1992. – Vol. 60, № 12. – P. 5139–5144.
© 2015 The Author(s). Published by All-Russian Scientific Research Institute of Fundamental and Applied Parasitology of Animals and Plants named after K.I. Skryabin.
This is an open access article under the Agreement of 02.07.2014 (Russian Science Citation Index (RSCI)) and the Agreemnt of 12/06/2014 (CABI org / Human Sciences section)